Koppelingsanalyse
Om er zeker van te zijn dat ons genetisch verhaal ook voor de
minder genetisch onderbouwden helder is, volgt hieronder een korte bespreking
over wat nu precies een LOD-score is. Het betreft hier een lichte bewerking van
de tekst die wij zelf ook gebruikten om dit fenomeen te doorgronden. Omwille van
de uitermate sterke didactische opbouw hebben wij er niet teveel aan gesleuteld
en enkel hier en daar wat anders bewoord en ingekort.
Inleiding
Wanneer
genetische kenmerken louter afhankelijk zijn van één locus dan is deze
mendeliaans. Hierbij komen we dan tot de overervingsmogelijkheden autosomaal
dominant (AD), autosomaal recessief (AR), geslachts(X)-gebonden dominant of
geslachts(X)-gebonden recessief.
Indien een genetisch kenmerk niet mendeliaans is, dan is deze multifactorieel,
oftewel bepaald door een combinatie van genetische en omgevingsfactoren en hoeft
dus niet door een enkele locus bepaald te worden.
Met behulp van koppelingsanalyse kan men kwantitatief bepalen of een kenmerk
bepaald wordt door één enkele locus die in de nabijheid ligt van andere reeds
gekende loci op een genoom en tevens de afstand hiertoe. Zo kan men dan het
kenmerk op de genetische kaart van het genoom zetten
Enkele definities en principes
locus:
fysische plaats
op een chromosoom. Op een locus bevindt zich een bepaalde sequentie of gen. Een
locus draagt een specifiek allel van dit gen.
allel:
een van de
alternatieve vormen van een gen op een bepaald locus.
genetische marker:
elk locus kan gebruikt worden als genetische marker van zodra het mogelijk is
genetische variatie ter hoogte van dit locus te detecteren.
haplotype:
in een strikte
betekenis, de specifieke allelen van verschillende loci die zich op dezelfde
fysische chromosoom bevinden. In de brede betekenis zeggen we dat een set
allelen voor verschillende loci een haplotype vormen. We erven dan een haplotype
van onze moeder en een haplotype van onze vader.
genotype:
genetische constitutie van een organisme. Ons genotype wordt gedefinieerd door
twee specifieke allelen voor elke locus.
Normaalgesproken is er sprake van onafhankelijke segregatie van allelen, dus
onafhankelijke overerving. Voor twee markers met als allelen respectievelijk A,
a en B, b zijn de mogelijke haplotypes van de gameten: AB, Ab, ab en aB. Deze
worden in een ratio 1:1:1:1 doorgegeven aan de nakomelingen.
Sommige paren van genen worden niet onafhankelijk overgeërfd, deze genen zijn
gekoppeld. Veronderstellen we een individu met als haplotypes AB en ab voor twee
loci zoals hierboven beschreven. Erven nakomelingen van dit individu het AB of
het ab haplotype, dan noemen we deze niet-recombinant voor deze
twee loci. Nakomelingen die een Ab of aB haplotype erven van deze ouder hebben
een recombinant haplotype. Segregeren deze twee genen niet
onafhankelijk, dan zullen de niet-recombinante haplotypes voor deze twee genen
frequenter voorkomen bij de nakomelingen dan de recombinante haplotypes. De
allelen van beide genen overgeërfd van één ouder zijn dan schijnbaar gekoppeld,
zij het niet absoluut. Dit fenomeen heet genetische koppeling
(genetische linkage). Kwantitatief wordt koppeling gemeten als de
recombinatiefractie
q,
de verhouding recombinanten/niet-recombinanten. Dit is ook de waarschijnlijkheid
dat een bepaalde ouder een recombinant haplotype doorgeeft aan een kind. Loci
die onafhankelijk overerven zijn niet gekoppeld. De geobserveerde
recombinatiefractie is dan
q =½.
Zijn twee loci volledig gekoppeld, dan komen er geen recombinanten
voor en q = 0.
Deze definitie van koppelingsanalyse heeft drie belangrijke gevolgen:
a) koppelingsanalyse vereist onderzoek van verwante individuen, niet verwante
individuen zullen geen informatie opleveren.
b) recombinante en niet-recombinante haplotypes kunnen niet altijd onderscheiden
worden. Stel dat een persoon een Ab/ab genotype bezit (de twee haplotypes zijn
dan Ab en ab). Omwille van de homozygositeit van de tweede locus (genotype b/b)
kan men de recombinante haplotypes afkomstig van dit individu niet onderscheiden
van de niet-recombinante haplotypes. Om dit onderscheid te kunnen maken moet een
persoon dus heterozygoot zijn voor beide loci. Dan alleen is een individu
informatief voor koppelingsanalyse.
c) een Ab haplotype is recombinant als het afkomstig is van een AB/ab individu
en niet-recombinant als het voortkomt van een individu met Ab/aB genotype. Deze
twee mogelijkheden voor een dubbele heterozygoot moeten dus onderscheiden worden
(met andere woorden, een persoon met haplotypes Ab en aB moet onderscheiden
worden van een persoon met haplotypes AB en ab). De specifieke haplotypes die
voorkomen bij een dubbele heterozygoot worden ook fase genoemd.
Homologe recombinatie
Gameten ontstaan
uit diploïde kiemcellen door meiose. Tijdens dit proces kan genetisch materiaal
uitgewisseld worden door homologe recombinatie of crossing-over. Zo
worden dus de recombinante haplotypes gegenereerd. Dergelijke crossing-over kan
overal op een chromosoom plaatsgrijpen. De kans dat dit voorkomt tussen twee
loci die op één chromosoom liggen is dus veel kleiner als deze dicht bijeen
liggen dan wanneer zij ver van elkaar verwijderd zijn. Er is dus een verband
tussen de fysische en de genetische afstand die twee loci scheidt. De
recombinatie fractie
q
wordt ook uitgedrukt in
centiMorgan (cM): 1 cM komt overeen met 1% recombinatie. Gemiddeld komt 1%
recombinatie overeen met 106 bp (1 mb). Hier kunnen lokaal echter
grote verschillen optreden: op specifieke plaatsen van het menselijk genoom kan
1% recombinatie overeenstemmen met 5 . 104 bp, op andere met meerdere
mb.
In het algemeen is er meer recombinatie aan de uiteinden van de chromosomen dan
rond de centromeer. Recombinatie is ook frequenter in de vrouwelijke meiose dan
in de mannelijke meiose. Er zijn dus vrouwelijke en mannelijke genetische
kaarten. Voor de meeste toepassingen wordt hiervan een gemiddelde genomen.
Kwantitatieve methodes
Het
hoofdprobleem bij de constructie van een genetische kaart van de mens is de
bepaling van de meest waarschijnlijke reële recombinatiefractie uitgaande
van een beperkt aantal observaties in families. Stel dat we binnen één
familie de overerving van twee merkers analyseren en een aantal recombinaties
waarnemen. De vraag is nu wat deze waarneming ons leert over de reële
recombinatiefrequentie tussen de twee merkers, en met welke statistische
zekerheid. Het is immers mogelijk dat de gemeten recombinatie frequentie in de
familie afwijkt van gemiddelde 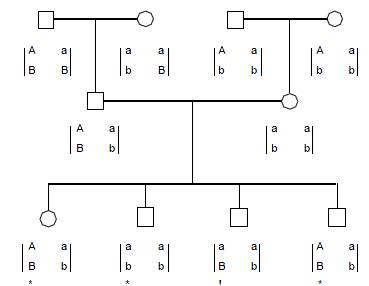 recombinatie
frequentie tussen de twee merkers in de populatie. Beschouwen we de overerving
van twee merkers de 3-generatie familie uit nevenstaande
stamboom. De vader
is dubbel heterozygoot voor beide merkers en dus informatief. De haplotypes van
de vader (of de
fase) kunnen bepaald
worden door de genotypes van zijn ouders te analyseren. De moeder is homozygoot
voor beide merkers en dus niet informatief. Er zijn
dus vier
informatieve chromosomen (preciezer: meioses), drie niet-recombinanten (*)
en één recombinant (!). De geobserveerde recombinatiefrequentie
tussen de merkers A en B binnen deze familie is dus 0,25. Het is echter
duidelijk dat als wij de hele populatie zouden onderzoeken de gemiddelde
recombinatiefrequentie tussen de twee merkers dan merkelijk zou kunnen
verschillen van de waarde in die familie, omdat recombinatie een toevallig
proces is.
recombinatie
frequentie tussen de twee merkers in de populatie. Beschouwen we de overerving
van twee merkers de 3-generatie familie uit nevenstaande
stamboom. De vader
is dubbel heterozygoot voor beide merkers en dus informatief. De haplotypes van
de vader (of de
fase) kunnen bepaald
worden door de genotypes van zijn ouders te analyseren. De moeder is homozygoot
voor beide merkers en dus niet informatief. Er zijn
dus vier
informatieve chromosomen (preciezer: meioses), drie niet-recombinanten (*)
en één recombinant (!). De geobserveerde recombinatiefrequentie
tussen de merkers A en B binnen deze familie is dus 0,25. Het is echter
duidelijk dat als wij de hele populatie zouden onderzoeken de gemiddelde
recombinatiefrequentie tussen de twee merkers dan merkelijk zou kunnen
verschillen van de waarde in die familie, omdat recombinatie een toevallig
proces is.
Er is dus behoefte aan een
statistische methode om, uitgaande van deze experimentele
waarnemingen, de gemiddelde
of reële recombinatiefractie te bepalen tussen merkers, en een idee te
krijgen over de statistische zekerheid waarmee wij die uitspraak kunnen doen. De
meest gebruikte statistische methode voor koppelingsanalyse is de lod score
(logarithm of odds) methode. L(q)
wordt gedefinieerd als de probabiliteit dat we een waarneming doen in een
experiment (familie) als de reële gemiddelde recombinatiefractie
q
is (gekoppelde merkers). De
wijze waarop L(q)
wordt berekend is dus afhankelijk van de structuur van de familie die we
analyseren. De waarschijnlijkheid dat er bij één meiose recombinatie optreed is
q.
De waarschijnlijkheid dat erbij één meiose geen recombinatie optreed is dus (1-q).
In deze familie is zijn vier informatieve meioses. Bij één van deze vier meioses
trad er recombinatie tussen de merkers op. Voor deze waarneming is L(q)
= (1-q)3q
Vervangen wij in die
formule q
door 0,5
(recombinatiefractie 0,5 of 50% recombinatie zoals wij verwachten voor twee
merkers die onafhankelijk segregeren), dan verkrijgen we L(½).
Dat is dan de probabiliteit dat we die waarneming doen als de twee merkers
volledig onafhankelijk overgeërfd worden. Voor die waarneming binnen die familie
is L(0,5) = 1/16. Dit betekent dat zelfs als de twee merkers volledig
onafhankelijk zijn er 1 kans op 16 is dat er in een familie zoals hierboven
beschreven slechts 1 recombinatie wordt waargenomen.Beide probabiliteiten (L(q)
en L(½))
hebben een waarde tussen 0 en 1. Absoluut betekenen deze getallen echter niet
veel omdat ze sterk afhangen van het aantal geobserveerde haplotypes in de
familie.
Daarom definiëren we de
lod score Z(q)
als:
Z(q)=log
[ L(q)/L(0,5)]
Die
formule geeft dan een absolute maat voor hoeveel waarschijnlijker het is dat wij
een bepaalde waarneming doen in een familie (experiment) als de recombinatie
fractie q
zou zijn, dan als die 0,5
zou zijn.
Door q
te laten variëren tussen 0
en 0,5 kunnen we de maximale lod score en de meest waarschijnlijke
recombinatiefractie bepalen. Een maximale lod score >3 bij een
recombinatiefractie
q wordt als
significante evidentie voor koppeling met de recombinatie fractie
q
beschouwd. Dit betekent dat
een bepaalde waarneming in een familie duizend maal waarschijnlijker is bij
koppeling van twee merkers met een recombinatiefractie
q
dan bij onafhankelijke
segregatie van deze merkers. Een lod score van -2 wordt als significantiegrens
voor niet-koppeling genomen.
In het voorbeeld hier is:
Z(q)
= log[16(1-q)3q]
Dus voor
q
= 0 is Z(q)
= -∞
q = 0,05 is Z(q)
= - 0,164
q
= 0,1 is Z(q)
= 0,067
q
= 0,25 is Z(q)
= 0,227
q
= 0,35 is Z(q)
= 0,187
De
probabiliteit dat twee of meer onafhankelijke waarnemingen samen voorkomen is
het product van de probabiliteiten voor elke waarneming afzonderlijk. Lod scores
zijn logaritmes, lod scores voor twee markers bekomen in verschillende families
kunnen dus opgeteld worden. Bekomen we zo een score > 3 dan zijn beide markers
gekoppeld met een bepaalde maximale recombinatiefractie
q.
Bekomen we een score < -2 dan is koppeling bij die
q
uitgesloten. Merk op dat
van het ogenblik dat er één recombinant haplotype wordt opgemerkt, de lod score
bij q=0
noodzakelijk -∞
is of de probabiliteit voor
q
= 0 is 0.
In de praktijk zal men dus nieuwe waarnemingen blijven doen tot Z(q)
groter is dan 3. Is Z(q)
> 3 dan zegt men dat
de twee merkers gekoppeld zijn en de
q
waarbij Z(q)
maximaal is, geeft de afstand tussen de twee merkers. Is Z(q)
< -2, dan zegt men dat de koppeling van de twee merkers met een deze
q
uitgesloten is.
Nog enkele nuttige definities:
Genetische
kaart: een
genetische kaart is een verzameling geordende, gekoppelde merkers die verspreid
zijn over het volledige genoom. De afstand tussen de merkers wordt gemeten als %
recombinatie en uitgedrukt in cM. De genetische kaart van de mens omvat ongeveer
3000 cM.
Genetische
heterogeniteit:
als er verschillende
genen zijn die onafhankelijk van elkaar éénzelfde fenotype veroorzaken, dan
spreken we van genetische heterogeniteit.
Penetrantie:
soms
leidt de aanwezigheid van een allel niet altijd tot het fenotype dat hiermee
samenhangt. We spreken dan van onvolledige penetrantie. Als een individu
het allel bezit, vertoont het het fenotype, of niet. Als we spreken van
80% penetrantie, betekent dit dat 80 % van de individuen die het allel
bezitten ook het fenotype vertonen
Variabele
expressie: de
aanwezigheid van een allel bij verschillende individuen kan tot een
verschillende ‘ernst’ van het fenotype leiden. Meestal is dit het gevolg van het
effect van andere (modifier) genen.
Pleiotrope
effecten – allelism: soms
kunnen de verschillende allelen van een gen tot verschillende fenotypes leiden.
Zo kan bijvoorbeeld een null allel tot een ernstig complex klinisch syndroom
leiden en een missense puntmutatie in hetzelfde gen enkel tot mentale
retardatie. Dit noemen we pleiotropisme of allelisme.
 recombinatie
frequentie tussen de twee merkers in de populatie. Beschouwen we de overerving
van twee merkers de 3-generatie familie uit nevenstaande
stamboom. De vader
is dubbel heterozygoot voor beide merkers en dus informatief. De haplotypes van
de vader (of de
fase) kunnen bepaald
worden door de genotypes van zijn ouders te analyseren. De moeder is homozygoot
voor beide merkers en dus niet informatief. Er zijn
dus vier
informatieve chromosomen (preciezer: meioses), drie niet-recombinanten (*)
en één recombinant (!). De geobserveerde recombinatiefrequentie
tussen de merkers A en B binnen deze familie is dus 0,25. Het is echter
duidelijk dat als wij de hele populatie zouden onderzoeken de gemiddelde
recombinatiefrequentie tussen de twee merkers dan merkelijk zou kunnen
verschillen van de waarde in die familie, omdat recombinatie een toevallig
proces is.
recombinatie
frequentie tussen de twee merkers in de populatie. Beschouwen we de overerving
van twee merkers de 3-generatie familie uit nevenstaande
stamboom. De vader
is dubbel heterozygoot voor beide merkers en dus informatief. De haplotypes van
de vader (of de
fase) kunnen bepaald
worden door de genotypes van zijn ouders te analyseren. De moeder is homozygoot
voor beide merkers en dus niet informatief. Er zijn
dus vier
informatieve chromosomen (preciezer: meioses), drie niet-recombinanten (*)
en één recombinant (!). De geobserveerde recombinatiefrequentie
tussen de merkers A en B binnen deze familie is dus 0,25. Het is echter
duidelijk dat als wij de hele populatie zouden onderzoeken de gemiddelde
recombinatiefrequentie tussen de twee merkers dan merkelijk zou kunnen
verschillen van de waarde in die familie, omdat recombinatie een toevallig
proces is.